嗜热四膜虫 I 型自剪接内含子是第一个被鉴定为催化 RNA 的纯 RNA 系统。 RNA 需要精确折叠成特定的三维结构才能在从蛋白质翻译到剪接的生物过程中发挥作用,但似乎极易发生错误折叠。 与许多结构化 RNA 一样,四膜虫核酶通过多种折叠途径组装,其中许多途径最终进入非天然动力学陷阱。 在体外标准条件下,一小部分四膜虫核酶在 1 分钟内折叠到天然 (N) 状态,而其余部分形成一个长寿命的错误折叠中间体 (M),要在数小时乃至数天后重新折叠到 N 状态。 这种化学开关的结构机制仍然未知。
2022年9月6日,中国科学技术大学张凯铭与美国斯坦福大学Rhiju Das、Wah Chiu教授团队合作在美国科学院院刊(PNAS)以直投方式(Direct submission)在线发表了冷冻电镜解析纯RNA三维结构领域的重要研究成果。 这是继2021年8月全长四膜虫核酶冷冻电镜结构被解析(Nature 2021)后,张凯铭团队在该领域的又一重要研究突破。

利用冷冻电镜解析高异质性和柔性样品结构的优势,研究人员解析了四膜虫 L-21 ScaI 核酶的错误折叠结构。从单个标本中获得了3种M亚态(M1、M2、M3)和1种N的冷冻电镜结构,总体分辨率分别为3.5 Å、3.8 Å、4.0 Å和3.0 Å。结构比较表明,除了包含核酶鸟苷结合位点的核心螺旋P7的旋转,以及连接P7与核酶核心其他元件的链J7/3和J8/7的交叉,M 亚态都与 N 高度相似(图一)。
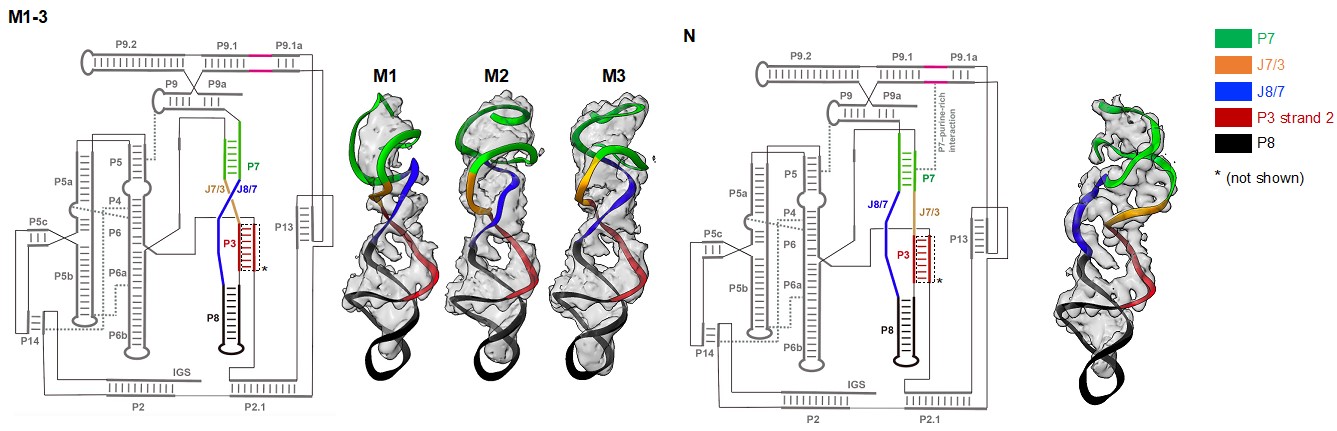
图一:M和N拓扑结构的不同。
M 亚态和 N之间的这种拓扑差异解释了M中5 ' -剪接位点底物对接失败的原因,支持了由于受困链交叉导致M向N缓慢重新折叠的“拓扑异构体”模型,并提出了M向N重新折叠的途径(图二)。
在从 M 到 N 的转变过程中,J7/3 和 J8/7 的交叉使得从 M 到 N 的直接转变在立体化学上是不可能的 (①)。 一个可能的 M-to-N 重折叠途径涉及 P3 和外周三级作用位点(例如 L9-P5、P13、P14)的瞬时断裂,这可以受许多因素的调节(②)。这些瞬时断裂将允许 P8 旋转,使 J7/3 和 J8/7 进入正确位置 (③)。 P3 重组和 P7 旋转将使 P7 与 P9.1 螺旋靠近,形成 P7多嘌呤相互作用并实现天然拓扑结构 (④)。

图二: 一种可能的M-to-N重折叠途径。在此示意图中,螺旋显示为圆柱体。外围半透明区是指“外围三级结构的松动”。
总的来说,本工作中,科研人员确认了M 和 N 之间存在的拓扑差异,并确定了交叉点涉及 J7/3 和 J8/7 核心链。 由于 J7/3 在前人的模型中没有被视为潜在的可移动核心链,因此在之前的研究中没有预料到这种特定的交叉,这说明了冷冻电镜分析在揭示重要结构特征方面的重要性和实用性。拓扑异构体可能广泛存在于复杂 RNA 的错误折叠中,这些冷冻电镜结构为剖析范式模型系统中的详细动力学机制和后续功能研究奠定了基础。
该研究获得了中国科学技术大学冷冻电镜平台和细胞动力学教育部重点实验的大力支持。中国科大李珊珊副研究员为本文第一作者;中国科大张凯铭教授、美国斯坦福大学Rhiju Das、Wah Chiu教授为本文通讯作者。该项研究获得中国科大科研启动经费和中科院率先行动引才计划择优经费的资助。
原文链接:https://www.pnas.org/doi/10.1073/pnas.2209146119
(生命科学与医学部、细胞动力学重点实验室、冷冻电镜平台、科研部)
